SARS-CoV2 besteht aus verschiedenen Virussträngen, auch Varianten genannt. Sie unterscheiden sich von der Ursprungsvariante (“Wildtyp” oder “Wuhan-Stamm”) durch eine Reihe von Mutationen im Genom, die das Virus infektiöser machen oder zur Immunflucht beitragen. Beides zusammen erhöht die Kontagiösität von SARS-CoV2. Unabhängig von der Variante heißt die Erkrankung aber weiterhin Covid19 und löst ähnliche Symptome sowie Folgeerkrankungen aus.
Folgende Informationen sind für Laien unerheblich: Sie ändern nichts daran, dass man sich mit einer Auffrischimpfung gegen schwere Verläufe schützen kann – bestenfalls mit dem angepassten Impfstoff, aber jeder Impfstoff ist besser als gar kein Impfstoff.
Die hier gesammelten Informationen sind aus persönlichem Interesse entstanden und mit Sicherheit unvollständig.
Hintergrund
- GISAID-Nomenklatur: Global Initiative on Sharing all Influenza Data, seit 2008 – Untervarianten werden in Kladen (geschlossene Abstammungsgemeinschaft) zusammengefasst, nach dem Einbuchstabencode mit der relevantesten Aminosäurenveränderung, z.B. Alpha als GRY (D614G, G204R und N501Y)
- Nextstrain: seit 2015 Echtzeitdaten, ebenfalls Kladen
- Pango-Nomenklatur: seit 2020, Darstellung als phylogenetischer Baum, feinerer Maßstab als Nextstrain und GISAID; Jede Linie hat eine numerische Bezeichnung, die mit A oder B beginnt. Jedes Mal, wenn sie einen Nachfolger bekommen, erhalten sie denselben Buchstaben mit einer neuen Zahl am Ende. Wenn B.1 weitere Nachfahren bekam, waren es B.1.1, B.1.2, etc… Damit die Zahlenreihen nicht zu ausschweifend wurden, zog man einen Deckel bei 3 Zahlen. Sobald eine Linie eine vierte Zahl bekam, wurden sie zum nächstverfügbaren Buchstaben im Alphabet konvertiert. Rekombinationen beginnen mit X.
- seit 31. Mai 2021 durch WHO: Buchstaben des griechischen Alphabets für Variants of Interest/Concern, seit 2022 nurmehr für Variants of Concern (Definitionen hier, Stand Oktober 2023)
- Mehr zum Varianten-Tracking der WHO hier, sowie von Cornelius Römer
Evolution von Alpha-Coronaviren, Beta-Coronaviren und Influenza H3N2
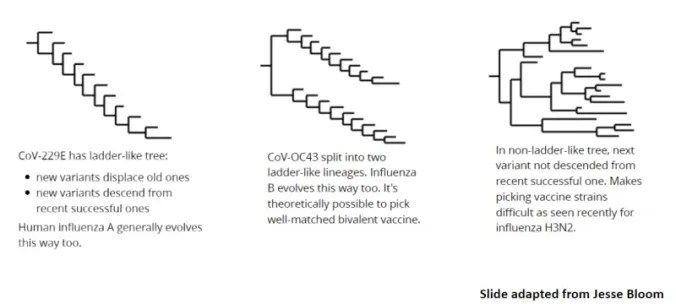
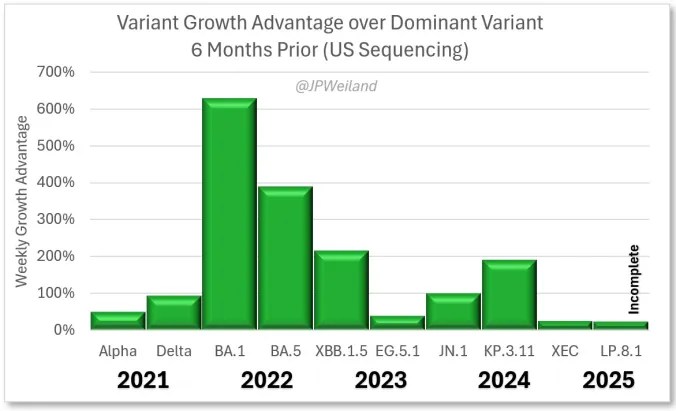
Die von Virologe Drosten pointiert formulierte “virologische Sackgasse” (Die Zeit, 23.11.22) ist eingetreten, wenn auch anders als ich Laie lange Zeit geglaubt habe. Natürlich entwickelt sich SARS-CoV2 stetig weiter, verhält sich wie eine Driftvariante, was eine zeitnahe Impfstoffentwicklung erschwert (mehr zum evolutionären Druck bei SARS-CoV2 von Adam Kucharski, 16.08.23). Es kommt zu kleineren und größeren Infektionswellen wie mit JN.1 (Herbst 2023) oder XFG (Herbst 2025), doch mit der steigenden Immunität in der Weltbevölkerung sinkt die Zahl der schweren Akutverläufe weiter stetig ab. Trotz teils beträchtlicher Anzahl an Mutationen kann das Virus unserem Immunsystem immer schlechter entkommen. Evolutionäre Sprünge mit dramatisch hohen Wellen sind seit JN.1 nicht mehr aufgetreten, bzw. entwickeln sich wesentlich langsamer (XFG, BA.3.2)

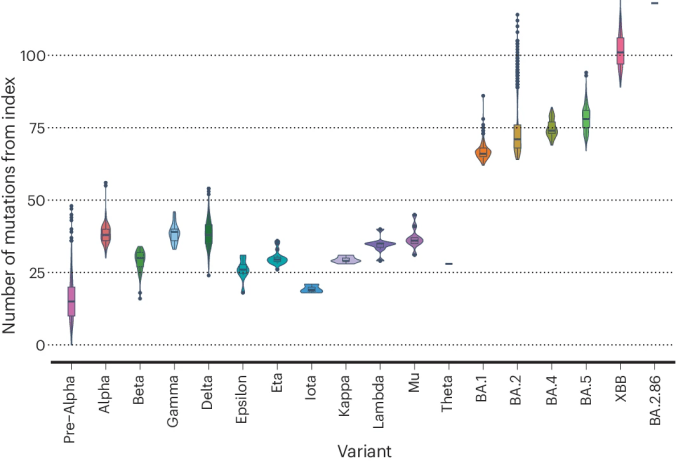
Omicron war ein „shift“-Ereignis, das der vorherrschenden Immunität entkam und neuen antigenischen Raum ausbeutete.
Relative und absolute Häufigkeit
Relative Häufigkeit ist der Anteil an allen Sequenzen zu einem bestimmten Zeitpunkt mit einer bestimmten Variante. Mit Jahresbeginn 2024 waren 70% aller sequenzierten Samples JN.1. Die absolute Häufigkeit ist die tatsächliche Zahl an Infektionen mit einer bestimmten Variante. Diese Zahl ist unklar, weil nicht jedes Sample sequenziert wird, weil viele Fälle nicht gemeldet werden. Wir kennen den Nenner nicht. Abwasserdaten umgehen dieses Problem teilweise, weil es keine Rolle spielt, ob Leute testen. Wir kennen dadurch die absolute Virusmenge, die ausgeschieden wird, und wie sie sich zusammensetzt.
Delta wurde komplett durch BA.1 ersetzt. Obwohl BA.2 ab März in den USA relativ dominant wurde, kann nicht die Rede davon sein, dass BA.1 komplett ersetzt wurde. Bei BA.2.86 nahmen selbst die Anteile der Nicht-BA.2.86-Sequenzen (v.a. HV.1.) während der JN.1.-Welle noch zu.
Das bedeutet, dass man die Zunahme von z.b. Hospitalisierungen nicht zwingend einer bestimmten Variante zuordnen kann.
Overshoot

Bis zur BA.2-Welle wurden Peak und Abfall der Welle in Österreich maßgeblich durch Schutzmaßnahmen sowie im Fall der Alpha-Welle Durchimpfungen beeinflusst. Die BA.5-Sommerwelle lief ungebremst durch, ebenso die hohe Frühwinterwelle mit JN.1 Ende 2023. Verantwortlich für den steilen Abfall nach dem Peak der JN.1-Welle war ein sogenannter Overshoot. Die Weihnachtsferien und die beginnende Influenzawelle können ebenfalls beigetragen haben, waren aber nicht hauptverantwortlich.
Der Peak ist dort, wo effektive Reproduktionszahl ca. 1 geworden ist, also der Herdenimmunitätssschwellenwert (HIT). R_eff ergibt sich aus der Reproduktionszahl vor Immunität und der Immunität der Bevölkerung:
R_eff = R0* (1-S), wobei S der Anteil der empfänglichen Personen ist.
Alle, die sich nach dem Overshoot anstecken, sind über HIT hinausgeschossen. Das drückt R_eff dann aber weiter, weil S deutlich sinkt.
Beeinflussungen der Höhe von Infektionswellen durch NPI:
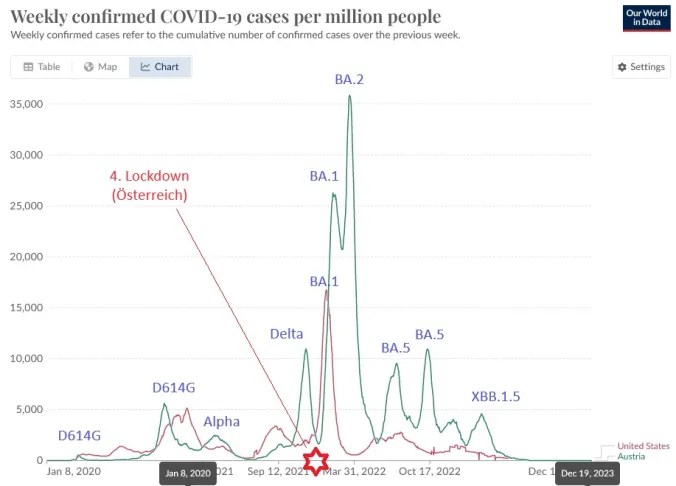
Die Delta-Welle im Spätherbst 2021 wurde durch den vierten Lockdown in Österreich abgeflacht, dann kamen noch die Weihnachtsferien. Das hat die BA.1-Welle deutlich gebremst. In den USA war die Delta-Welle deutlich geringer, dafür gab es nur eine BA.1-Welle. Diesen Doppelgipfel von BA.1 und BA.2 als langgestreckte Welle gab es imho nur in Mitteleuropa. Die Alpha-Welle wiederum wurde durch die Impfkampagne im Frühjahr 2021 deutlich abgeflacht und bremste den Anstieg der Delta-Welle. Die kurzen Wellenabstände im ersten Omicron-Jahr bis XBB.1.5 stammten wahrscheinlich auch von der mangelnden Antikörperdiversität (s.o.) – für viele Menschen war es die erste Infektion.
Laufende Übersicht
(Stand November 2025)
Übersicht über prominentere Varianten
- Wildtyp (A und B)
- D614G-Variante (seit Februar 2020, höhere Infektiösität)
- Alpha (B.1.1.7, N501Y), infektiöser als die D614G-Variante
- Beta (B.1.351, N501Y, E484K), mehr Immun Escape
- Gamma (P.1, N501Y, E484K, K417T), infektiöser, vermehrt Reinfektionen (Yépez et al. 2022)
- Delta (B.1.1.617.2, P681R, L452R, T478K, siehe Dhawan et al. 2022 ), infektiöser als Alpha; Delta plus variant war noch tödlicher (mit K417N, V70F, W258L)
- Omicron (B.1.1.529, 66 Mutationen, von Beginn an BA.1 und BA.2), viel mehr Immun Escape
- BA.1, weniger pathogen als Delta, geringere Viruskonzentration im Stuhl (Baldovin et al. 2023)
- BA.2 (ohne Löschung 69/70), pathogener als BA.1
- BA.4/BA.5, vermutlich Rekombination
- XBB.1.5 (Rekombinante, F486P)
- EG.5.1 (F456L, Q52H), VOI
- HV.1 (S:F456L+L452R)
- Pirola (BA.2.86, über 30 Mutationen, P681R), keine Rekombinante, vmtl. im Sommer 2022 entstanden durch chronische Infektion, VOI seit 22.11.23
- JN.1 (L455S, separate VOI seit 18.12.23, signifikanter Immun Escape, besser übertragbar, keine leichteren Verläufe)
- KP.2 (JN.1 + F456L, R346T und V1104L) und KP.3 (ohne R346T, mit Q493E)
- KP.3.1.1. (mit S31Del), signifikant höhere ACE2-Bindung und höherer Immun Escape;
- XEC (Rekombination aus KS.1.1 und KP.3.3., zusätzlich noch T22N, F59S – , Ende Juni erstmals in Berlin detektiert, „EM-Variante“, mutiertes N*-Protein obsolet), sorgte für kleinere Welle im Dezember 2024
- LP.8.1. (LP.8 fügt Q493E hinzu), hat R190S (Glykane), zudem S31del, Wachstumsvorteil gegenüber XEC, vor allem deutlich mehr ACE2-Bindung, infektiöser (seit 24.01.25 Variant of Monitoring)
- NB.1.8.1 – konvergente Variante, Schlüsselmutation A435S (starker Immun Escape – bereits beim Wildtyp nachgewiesen Li et al. 2020, hohe ACE2-Bindung erhalten – Guo et al. 2025 preprint, seit Mai eine VOM)
- XFG – Rekombinante, hohe weltweite Dominanz im Herbst 2025
- BA.3.2 und Nachfolge-Varianten könnten 2026 neue Wellen auslösen (Stand Oktober v.a. in Australien aktiv)
Addendum
Folgende Texte gehen schon sehr ins Detail und ist wirklich nur etwas für Nerds. Auch hier gilt, dass ich wesentlich mehr Information zusammengetragen als verstanden habe.
Kryptische Linien
Mutationsspielräume
Varianten-Steckbriefe
Ich hab die unterschiedlichen Varianten bis XFG und NB.1.8.1 (Herbst 2025) verfolgt. Danach ergaben sich für mich keine bahnbrechend neuen Erkenntnisse mehr, außer, dass SARS-CoV2 niemals ausmutiert haben wird. Sollte es wieder einmal zu einem evolutionären Sprung kommen, knüpfe ich hier an und werde berichten – sofern es noch Surveillance-Daten gibt. Sonst kann man davon ausgehen, dass weiterhin SARS-CoV2 zirkuliert und die Erkrankung Covid19 verursacht – mit welcher Variante auch immer, und der aktuelle Impfstoff der beste Impfstoff sein wird.
You must be logged in to post a comment.